1. harmoic reg(조화 회귀)이란?
https://pastryofjsmath.tistory.com/91
[R] AirPassengers 데이터를 이용한 회귀분석 비교(회귀분석,동적회귀)
위 데이터를 다루는데 있어 어떠한 모형인지부터 알고 시작하자.data('AirPassengers')AP AP Jan Feb Mar Apr May Jun Jul Aug Sep Oct Nov Dec1949 112 118 132 129 121 135 148 148 136 119 104 1181950 115 126 141 135 125 149 170 170 158 133
pastryofjsmath.tistory.com
2. 푸리에급수란?
https://pastryofjsmath.tistory.com/92
LA21) 푸리에급수(Fourier series)-1
이번 파트에서는 푸리에급수를 다루려고 한다.갑자기 벡터를 다루더니 왠 급수를 다루냐?? 라고 할 수 있다. 하지만 다항식도 선형대수에서 다루는 영역 중 하나이고기저 다항식들을 기반으로
pastryofjsmath.tistory.com
3. 데이터
공공데이터 포털에서 서울시 미세먼지에 대한 데이터를 가져옴.
미세먼지에 영향이 있는 여러 요인들을 더 탐색해서 하고 싶었으나 시간상 여유가 안되어서 이렇게 진행함.
https://www.data.go.kr/data/15089266/fileData.do
서울특별시_시간별 (초)미세먼지_20221231
서울특별시 대기질 자료(초미세먼지, 미세먼지) 입니다.<br/>2008년 1월부터 2022년 12월 31일까지의 자료로 자치구별 시간 평균 자료(서울시 평균 자료 포함)입니다.<br/>본 측정자료는 확정 전 실시
www.data.go.kr
4. 데이터 전처리,시각화
rm(list=ls())
gc()
data <- read.csv('C:/Users/PJS/Desktop/R 데이터/서울특별시_시간별 (초)미세먼지_20221231/서울시 대기질 자료 제공_2008-2011.csv',header = T,fileEncoding = 'CP949')
#library(dplyr)
#data %>% View()
data$구분 <- as.factor(data$구분)
data %>% View()
#library(ggplot2)
#ggplot은 필터링된 데이터를 직접 참조가 안됨.
#필터링은 둘 다 가능함 원하는 방식대로 사용
new_data <-data[(data$구분 %in% c('관악구','강동구','강남구','강서구','강북구')),]
new_data <- data %>% filter(구분 %in% c('관악구','강동구','강남구','강서구','강북구'))
#levels(new_data$구분)
#unique(new_data$구분)
#결측 데이터 체크
colSums(is.na(new_data))
na.omit(new_data)
rownames(new_data) <- 1:nrow(new_data)
데이터 수가 10만개가 넘어갑니다. 시간별 데이터로 되어있기에 일별,월별 등으로 전처리 과정이 필요합니다.
여기서는 group_by()를 이용하여 일별 데이터로 전처리 했습니다.
여기서 날짜 데이터 전처리 방법을 잘 모르시는분은
https://pastryofjsmath.tistory.com/35
날짜 데이터 전처리 [in R] -strptime,str_extract(),substr()
https://pastryofjsmath.tistory.com/28 날짜,시간 데이터 전처리*위의 파일을 이용하여 날짜와 시간 데이터 전처리하였습니다.import pandas as pdimport numpy as np#df_merged_mean = pd.read_csv('파일위치주소',encoding='UFT
pastryofjsmath.tistory.com
new_data$일시 <- strptime(x = new_data$일시,format= '%Y-%m-%d%H:%M')
new_data$날짜 <- as.Date(new_data$일시)
daily_summary_mean_pm25 <- new_data %>% group_by(날짜,구분) %>% summarise(평균_PM25 = mean(초미세먼지.PM25.,na.rm = T)) %>% ungroup()
daily_summary_mean_pm25
daily_summary_mean_pm25<-na.omit(daily_summary_mean_pm25)
str(daily_summary_mean_pm25)
> str(daily_summary_mean_pm25)
tibble [7,053 × 3] (S3: tbl_df/tbl/data.frame)
$ 날짜 : Date[1:7053], format: "2008-01-01" "2008-01-01" "2008-01-01" ...
$ 구분 : Factor w/ 26 levels "강남구","강동구",..: 2 3 4 5 2 3 4 5 2 3 ...
$ 평균_PM25: num [1:7053] 15.6 13.1 13.1 12 18.5 ...
- attr(*, "na.action")= 'omit' Named int [1:87] 233 238 279 284 3302 3307 3312 3368 3373 4568 ...
..- attr(*, "names")= chr [1:87] "233" "238" "279" "284" ...
ggplot(data=daily_summary_mean_pm25, aes(x = 날짜, y= 평균_PM25,color=구분))+
geom_line()+
theme_minimal()+
labs(title = "구별 일별 초미세먼지(PM2.5) 추이",
x = "날짜", y = "평균 PM2.5 (㎍/㎥)")+
ylim(0,200)
ggplot(daily_summary_mean_pm25, aes(x = 날짜, y = 평균_PM25)) +
geom_line(color = "steelblue") +
facet_wrap(~ 구분, ncol = 2) +
labs(title = "구별 일별 초미세먼지(PM2.5) 추이 (facet)",
x = "날짜", y = "평균 PM2.5 (㎍/㎥)") +
theme_minimal()+
ylim(0,200)
상당히 어지럽고 그래프의 추세선도 잘 보이지 않습니다. 그럴때는 다음과 같이 요인별로 분류하면 됩니다.

여기서 "관악구"를 예측해보겠습니다.
gwanakgu <- daily_summary_mean_pm25 %>% filter(구분 == '관악구')
N<- nrow(gwanakgu)
t <- 1:N
logy <- log(gwanakgu$평균_PM25)
gwanakgu$평균_PM25 <- logy
train_idx <- 1:round(0.7*N)
test_idx <- (round(0.7*N)+1) : N
train_df <- gwanakgu[train_idx,]
test_df <- gwanakgu[test_idx,]
t_train <- 1:nrow(train_df)
t_test <- (nrow(train_df)+1):(nrow(train_df)+nrow(test_df))
create_fourier_feat <- function(time,K,period){
cos_terms <- sapply(1:K, FUN = function(k) cos(2*pi*k*time/period))
sin_terms <- sapply(1:K, FUN = function(k) sin(2*pi*k*time/period))
fourier_feat <- cbind(cos_terms,sin_terms)
colnames(fourier_feat) <- c(paste0('cos',1:K),paste0('sin',1:K))
return(fourier_feat)
}반드시! 순서대로 t를 지정해주셔야 합니다. 시계열로 보겠다는 것이니까요.
#훈련데이터 fitting
fourier_train <- create_fourier_feat(t_train,K=3,period=7)
fourier_train_df <- cbind(y=train_df$평균_PM25,fourier_train)
fourier_test <- create_fourier_feat(t_test,K=3,period=7)
model_fourier <- lm(y~.,data=data.frame(fourier_train_df))
pred_log <- predict(model_fourier, newdata= as.data.frame(fourier_test))
pred <- exp(pred_log)
actual_y <- exp(test_df$평균_PM25)
mse_fourier <- mean((actual_y - pred)^2,na.rm = T)
mse_fourier
[1] 294.1781
# SSR: 예측값과 평균값 사이의 제곱합
ssr <- sum((pred - mean(actual_y))^2)
# SST: 실제값과 평균값 사이의 제곱합
sst <- sum((actual_y - mean(actual_y))^2)
# 테스트셋 기준 R²
r2_test <- ssr / sst
cat("테스트셋 기준 R²:", round(r2_test, 4), "\n")
테스트셋 기준 R²: 0.0377
df<-data.frame(pred=pred,y=exp(test_df$평균_PM25))
plot(gwanakgu$날짜,exp(gwanakgu$평균_PM25),type='l',ylim=c(0,50))
lines(test_df$날짜,pred,col='blue',lwd=1,lty=2)
legend("topleft", legend = c("실제값", "Fourier 회귀 예측"),
col = c("grey", "blue"), lty = c(1,2), lwd = 2)
처참한 수준입니다..
하지만 파라미터 k를 조절해서 더 타이트하게 만들 수 있습니다.
gwanakgu$log_PM25 <- log(gwanakgu$평균_PM25)
train_df <- gwanakgu[train_idx, ]
test_df <- gwanakgu[test_idx, ]
t_train <- 1:nrow(train_df)
t_test <- (nrow(train_df)+1):(nrow(train_df)+nrow(test_df))
#period와 K 조정
K <- 1200
period <- 7
fourier_train <- create_fourier_feat(t_train, K = K, period = period)
fourier_test <- create_fourier_feat(t_test, K = K, period = period)
model_fourier <- lm(log_PM25 ~ ., data = data.frame(log_PM25 = train_df$log_PM25, fourier_train))
pred_log <- predict(model_fourier, newdata = as.data.frame(fourier_test))
pred <- exp(pred_log)
actual_y <- test_df$평균_PM25
mean((actual_y - pred)^2,na.rm=T)
# 시각화
plot(gwanakgu$날짜, exp(gwanakgu$평균_PM25), type='l')
lines(test_df$날짜, exp(actual_y), col='blue', lwd=1, lty=2)
legend("topleft", legend = c("실제값", "Fourier 회귀 예측"),
col = c("grey", "blue"), lty = c(1,2), lwd = 2)
# SSR: 예측값과 평균값 사이의 제곱합
ssr <- sum((pred - mean(actual_y))^2)
# SST: 실제값과 평균값 사이의 제곱합
sst <- sum((actual_y - mean(actual_y))^2)
# 테스트셋 기준 R²
r2_test <- ssr / sst
cat("테스트셋 기준 R²:", round(r2_test, 4), "\n")
얼핏보기엔 잘 예측된다. 라고 볼 수 있지만
이것에 대한 설명력은 어떻게 될지는 아직 모릅니다.
K값을 늘려가면서 R2가 어떻게 변화하는지 시각적으로 보는 코드입니다.
오래걸리고 메모리를 많이 먹기에 주기적으로 아래 코드로 메모리를 비워주세요.
rm(list=ls())
gc()
for (i in seq_along(K_vals)) {
K <- K_vals[i]
ft_train <- create_fourier_feat(t_train, K, period = 7)
ft_test <- create_fourier_feat(t_test, K, period = 7)
model <- lm(log_PM25 ~ ., data = data.frame(log_PM25 = train_df$log_PM25, ft_train))
pred_log <- predict(model, newdata = as.data.frame(ft_test))
pred <- exp(pred_log)
ssr <- sum((pred - mean(actual_y))^2)
sst <- sum((actual_y - mean(actual_y))^2)
results$R2_test[i] <- ssr / sst
}
plot(results$K, results$R2_test, type = 'b',
main = "K에 따른 테스트셋 R² 변화",
xlab = "K", ylab = "R² (Test)")
저는 중간에 멈추었기에 1500까지만 그려졌습니다.
하지만 파라미터를 계속 키워서 확 과적합시켜버리면 의미가 없을테니까
하나의 지표 MSE를 R2와 비교해가면서 확인하면 될 것 같습니다.
K_vals <- seq(1200, 1500, by = 50)
results <- data.frame(K = K_vals, R2_test = NA, MSE_test = NA)
for (i in seq_along(K_vals)) {
K <- K_vals[i]
# Fourier 특성 생성
ft_train <- create_fourier_feat(t_train, K, period = 7)
ft_test <- create_fourier_feat(t_test, K, period = 7)
# 모델 학습
model <- lm(log_PM25 ~ ., data = data.frame(log_PM25 = train_df$log_PM25, ft_train))
pred_log <- predict(model, newdata = as.data.frame(ft_test))
pred <- exp(pred_log)
# 성능 지표 계산
actual_y <- test_df$평균_PM25
ssr <- sum((pred - mean(actual_y))^2, na.rm = TRUE)
sst <- sum((actual_y - mean(actual_y))^2, na.rm = TRUE)
r2_test <- ssr / sst
mse_test <- mean((actual_y - pred)^2, na.rm = TRUE)
results$R2_test[i] <- r2_test
results$MSE_test[i] <- mse_test
}
#R² & MSE 동시 시각화
plot(results$K, results$R2_test, type = 'b', col = "blue", lwd = 2,
xlab = "K", ylab = "R² (파랑) / MSE (빨강)",
main = "K 값에 따른 테스트셋 R²와 MSE 변화")
# 두 번째 y축: par(new=TRUE)
par(new = TRUE)
plot(results$K, results$MSE_test, type = 'b', col = "red", lwd = 2, axes = FALSE,
xlab = "", ylab = "")
axis(side = 4, col = "red", col.axis = "red")
mtext("MSE (빨강)", side = 4, line = 3, col = "red")
legend("bottomright", legend = c("R² (Test)", "MSE (Test)"),
col = c("blue", "red"), lty = 1, lwd = 2)
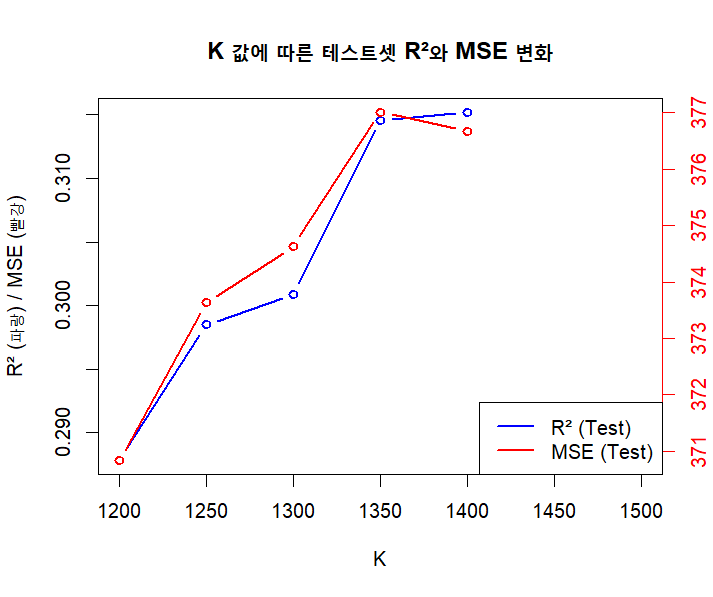
서로 값이 겹치는 구간이 존재하는데 저기 값을 파라미터로 쓰면 되겠습니다.
전체코드는 아래 깃허브에 올려두었습니다.
https://github.com/juseong4121/R
GitHub - juseong4121/R
Contribute to juseong4121/R development by creating an account on GitHub.
github.com